Génétique somatique
Génétique somatique :
Dans le cadre des missions diagnostiques, pronostiques, prédictives et épidémiologiques qui sont les nôtres, CYPATH a développé une plateforme de Génétique Somatique des cancers.
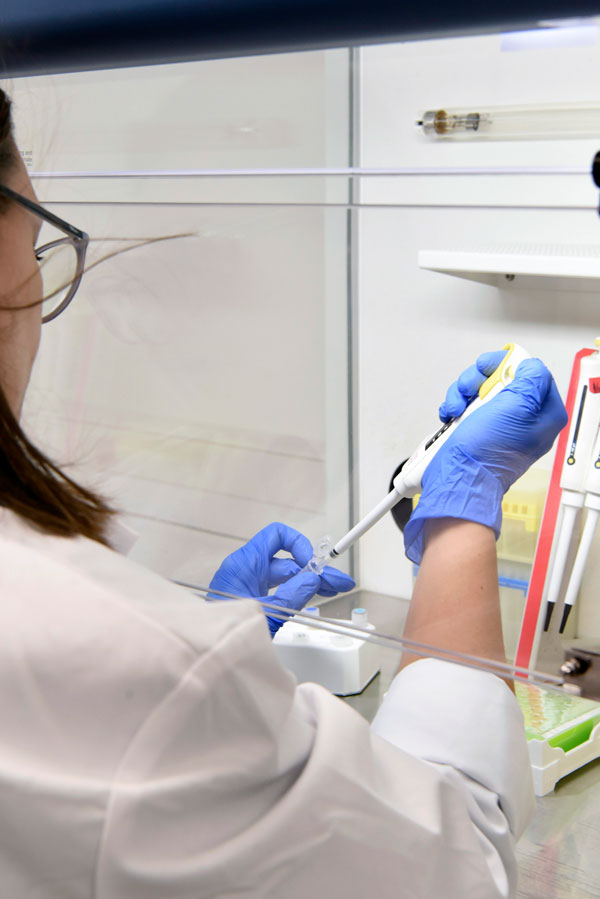
Techniques :
PCR temps réel :
Cette technique permet une détection qualitative des mutations à l’aide de cartouches utilisant des amorces spécifiques d’allèles.
- La cartouche BRAF Mutation Test permet la détection des mutations V600E/E2/D et V600K/R/M sur le codon 600 du gène BRAF dans les mélanomes.
- La cartouche EGFR Mutation Test permet la détection de 51 mutations des exons 18, 19, 20 et 21 du gène EGFR dans les cancers pulmonaires.
- La cartouche KRAS Mutation Test permet la détection de 21 mutations KRAS des exons 2, 3 et 4 dans les cancers du colon.
- La cartouche NRAS/BRAF Mutation Test permet la détection de 18 mutations NRAS des exons 2, 3 et 4 et 5 mutations BRAF dans le codon 600 dans les cancers du colon.
- La cartouche MSI Mutation Test permet la détection qualitative des mutations dans 7 nouveaux locus MSI permettant de déterminer si les tumeurs présentent une instabilité des microsatellites, reflet d’un défaut de système de réparation de l’ADN.
Séquençage Nouvelle Génération (NGS) :
Le séquençage nouvelle génération (NGS), ou séquençage à haut débit permet de séquencer rapidement l’ADN et l’ARN. A partir d’un prélèvement tissulaire parfois minime, il permet la détection d’un grand nombre de mutations sur un grand nombre de gènes avec une bonne sensibilité et un délai rapide.
La technique utilisée est la technique capture avec le séquenceur NextSeq 550DX Illumina, et des panels Agilent sur ADN pour tumeurs solides (39 gènes), sur ARN pour tumeurs solides (61 gènes), et sur ADN pour les gènes de recombinaison homologue (BRCA1, BRCA2 et 15 autres gènes).
Liste exhaustive des panels
- Panel NGS ADN sur tumeur solide :
AKT1 (NM_005163.2)(exons 03, 04), ALK (NM_004304.5) (exons 04, 05, 06, 20-29), BRAF (NM_004333.6) (exons 11,15), CDK4 (NM_000075.4) (exon 02), CDKN2A (NM_000077.4) (exon 01-03), CTNNB1 (NM_001904.4) (exons 03, 04), DDR2 (NM_006182.4) ( exons 03-18), EGFR (NM_005228.5) (exons 03, 06, 07, 09, 14, 15, 18, 19, 20, 21, 28), ERBB2 (NM_004448.3) (exons 08, 11, 12, 17, 19, 20, 21, 23, 24, 25), ERBB3 (NM_001982.3) (exons 03, 07, 08, 23), ERBB4 (NM_005235.3) (exons 10,12), FGFR1 (NM_023110.3) (exons 04, 07, 11, 12, 13, 14, 15), FGFR2 (NM_000141.4) (exons 04-15), FGFR3 (NM_000142.4) (exons 07, 09, 12, 14, 16), FOXL2 (NM_023067.4) (exon 01), GNAS (NM_000516.6) (exons 08, 09), H3F3A (NM_002107.6) (exon 02), HIST1H3B (NM_003537.3) (exon 01), HNF1A (NM_000545.6) (exons 01-06), HRAS (NM_005343.4) (exons 02-04), IDH1 (NM_005896.3) (exon 04), IDH2 (NM_002168.3) (exon 04), KEAP1 (NM_203500.2) (exon 03), KIT (NM_000222.2) (exons 08, 09, 10, 11, 13, 14, 17, 18), KRAS (NM_004985.5) (exons 02-04), MAP2K1 (NM_002755.3) (exons 02-03), MET (NM_000245.4) (exons 02, 09, 13-20), NOTCH1 (NM_017617.5) (exons 20, 26, 27), NRAS (NM_002524.5) (exons 02-04), PDGFRA (NM_006206.6) (exons 12, 14, 18), PIK3CA (NM_006218.4) (exons 02, 03, 06, 08, 10, 11, 21), PIK3KR1 (NM_181523.3) (exons 02, 03, 04, 05, 06, 07, 09, 11, 13), POLE (NM_006231.4) (exons 03, 19), PTEN (NM_000314.8) (exons 01-09), RB1 (NM_000321.2) (gène complet), ROS1 (NM_002944.2) (exons 38, 41), SMAD4 (NM_005359.6) (exons 03, 05, 08-12), STK11 (NM_000455.5) (exons 01-09), TERT (promoteur), TP53 (NM_000546.5) (exons 02-09). - Panel NGS ARN sur tumeur solide :
ACTB (NM_001101.5), AKT3 (NM_005465) (exons 01-03), ALK (NM_004304) (exons 02, 04, 06, 10, 16-23, 26 et intron 19), AXL (NM_021913) (exon 18, 19, 20), BRAF (NM_004333) (exons 01-03, 07-13, 15, 16), BRD8 (NM_139199.2), CAMTA1 (NM_015215) (exon 01-03), CCNB3 (NM_033031) (exons 02-06), CITED2 (NM_006079.5), COL6A3 (NM_004369.4), CREBBP (NM_004380.3), EGFR (NM_005228) (exons 01-09, 16, 19, 20, 24, 25), EMILIN2 (NM_032048.3), EP400 (NM_015409.5), EPC1 (NM_025209) (exons 09-11), EPC1 (NM_015630.4), ERG (NM_004449) (exons 02-11), ETV1 (NM_004956) (exon 03-13), ETV4 (NM_001986) (exon 02-10), ETV5 (NM_004454) (exon 02, 03, 07-09), EWSR1 (NM_005243) (exons 04-14), FGFR1 (NM_015850) (exon 02-12, 17), FGFR2 (NM_000141) (exon 02, 05, 07-10, 16, 17), FGFR3 (NM_000142) (exon 03, 05, 08-10,16-18, intron 17), FN1 (NM_212482.4), FOXO1 (NM_002015) (exon 01-03), FUS (NM_004960) (exon 04-11, 14), HSPA8 (NM_006597.6) , IRF2BP2 (NM_182972.2), JAZF1 (NM_175061) (exon 02-04), MALM2 (NM_032427.4) (exons 02-05), MEAF6 (NM_001270875) (exons 04, 05), MET (NM_000245) (exon 02-06, 13-17, 21), MYB (NM_001130173) (exon 05-16), NCOA2 (NM_006540) (exon 11-14), NR4A3 (NM_006981.4) (exon 03, 05-08), NRG1 (NM_004495) (exon 01-04, 06, 08), NTRK1 (NM_002529) (exons 02, 04, 06, 18, 10-13), NTRK2 (NM_006180) (exons 05, 07, 09, 1-17), NTRK3 (NM_002530) (exons 04, 07, 10, 12-16), PAX3 (NM181459.4), PDGFB (NM_002608) (exons 02, 03), PDGFRA (NM_006206.6) (exons 07-14), PLAG1 (NM_002655) (exons 01-04), PPARG (NM_015869) (exons 01-03, 05), RAD51B (NM_00133510.4), RAF1 (NM_002880) (exons 04-07, 09-10), RELA (NM_0021975) (exons 03, 04), RET (NM_0020630) (exons 02, 04, 06, 08-11), ROS (NM_002944) (exons 02, 04, 07, 31 à 37), SS18 (NM_001007559) (exons 04-06, 08-11), SS18L1 (NM_198935.3), TAF15 (NM_139215) (exons 05-07), TCF12 (NM_207036) (exons 04-06), TFE3 (NM_006521) (exons 02-08), TFG (NM_006070) (exons 04-07), VGLL2 (NM_153453.1), WWTR1 (NM_015472.6), YWHAE (NM_006761) (exons 05), ZC3H7B (NM_017590.6), ZFP36 (NM_003407.5). - Panel NGS ADN recombinaison homologue (cancers de l’ovaire, sein, pancréas, prostate) (étude Great) :
ARID1A (NM_006015.6), ATM (NM_000051.4), BRAF (NM_004333.6), BRCA1 (NM_007300.4), BRCA2 (NM_000059.4), BRIP1 (NM_032043.3), CDK12 (NM_016507.4), CHEK1 (NM_001274.5), CHEK2 (NM_007194.4), FANCA (NM_000135.4), FANCL (NM_018062.4), NBN (NM_002485.5), PALB2 (NM_024675.4), PIK3CA (NM_006218.4), RAD51C (NM_058216.3), RAD51D (NM_002878.4), TP53 (NM_000546.5).
Panels par organe (liste non-exhaustive)
- Cancer du poumon :
Mutations de EGFR KRAS BRAF HER2 MET STK11
Réarrangements de ALK ROS MET NTRK1 NTRK2 NTRK3 RET NRG1 - Cancer du sein :
Mutations de HER2 PIK3CA POLE
Réarrangements de ETV6-NTRK3 (diagnostic de carcinome sécrétoire) - Endomètre :
Mutations de PTEN PIK3CA POLE - Tumeurs de la granulosa (diagnostic) :
Mutations de FOXL2 - Mélanome :
Mutations de BRAF NRAS KIT HRAS
Réarrangements de ALK ROS RET MET NTRK1 NTRK3 - Rein :
Réarrangements de TFE3 ALK - Vessie :
Mutations FGFR3 FGFR2 HER2 PIK3CA POLE
Réarrangements de FGFR2 FGFR3 - Thyroïde :
Mutations de BRAF NRAS HRAS - Cancer colo-rectal :
Mutations de KRAS NRAS BRAF - Pancréas :
Mutations de KRAS P53 SMAD4 CDKN2A
Réarrangements de NRG1 NTRK - Cholangiocarcinome :
Mutations IDH1 IDH2 BRAF HER2 PIK3CA - Adénomes hépatiques (diagnostic) :
Mutations de HNF1 GNAS CTNNB1 - GIST :
Mutations de KIT PDGFRA - Tumeurs conjonctives (diagnostic) :
Fibromatose desmoïde : mutations de CTNNB1 ; Dermatofibrosarcome protuberans : réarrangements de COL1A1-PDGFB, COL6A3/EMILIN2-PDGFD ; Synovialosarcome : réarrangements de SS18/SS18L1-SSX ; Liposarcome myxoïde : EWSR1/FUS-DDIT3 ; Sarcome d’Ewing : FUS-FEV/ERG, EWSR1-FLI1/ETV1/FEV/E1AF/SMARCA5/POU5F1/SP3 ; Hemangioendotheliome épithélioïde : WWTR1-CAMTA1, YAP1-TFE3 ; Sarcome fibromyxoïde bas grade : EWSR1-CREB3L1/L2, FUS-CREB3L1/L2 ; Sarcome du stroma endométrial de bas grade : réarrangements de JAZF1-PHF1/SUZ12, … - Gènes du système de recombinaison homologue (BRCA1/2 etc.…) :
Cancer de l’ovaire, sein, prostate, pancréas
Sensibilité analytique 5%.
Après une étape de bioinformatique/bioanalyse, les mutations sont interprétées selon les connaissances médicales par une équipe de médecins moléculaires.
Signature génomique dans le cancer du sein :
Le test EndoPredict de Myriad International est un test qualitatif qui mesure l’expression de 12 gènes différents et utilise la technologie de la réaction en chaîne par polymérase quantitative par transcription inverse (RT-qPCR).
Il établit un risque de récidive à distance chez les patientes présentant un cancer du sein primaire à récepteurs d’œstrogènes positifs, HER2 négatif et traitées exclusivement par hormonothérapie adjuvante.
Le score moléculaire de 12 gènes, associé aux données cliniques relatives à la taille de la tumeur et au nombre de ganglions lymphatiques positifs, permet de calculer le Score EPclin plus significatif.
La « valeur seuil » permet de classer l’échantillon en fonction du risque élevé ou faible de récidive à distance.
Hybridation in situ en fluorescence (FISH) :
La FISH a été mise au point au cabinet Cypath pour le gène cerbB2 (protéine HER2) dans le cancer du sein pour l’indication d’une thérapie ciblée (Herceptin), depuis 3 ans, comme la technique gold standard.
Cette technique doit maintenant être étendue à d’autres zones d’intérêt pouvant servir à des diagnostics de sarcomes (MDM2 et autres), lymphomes (BCL2, BCL6, cMyc), tumeurs rares et à l’indication de traitements ciblés dans le cancer du poumon (ALK, ROS1, MET).

Qualité :
Le cabinet Cypath est accrédité en génétique somatique par le COFRAC selon la norme ISO 15 189 depuis 2017. Cette accréditation reflète la mise en place d’une organisation garantissant des pratiques totalement maîtrisées et contrôlées (évaluations externes de la qualité). Il s’agit également d’une condition indispensable à la facturation de certains actes (liste complémentaire du RIHN).
Prise en charge financière :
Cette activité est financée, dans le cadre de la législation actuelle, selon le référentiel des actes innovants (RIHN) pour que les patients puissent bénéficier de ces tests moléculaires non-inscrits à ce jour à la nomenclature.
Nous avons développé un partenariat actif avec l’ensemble des établissements de soins avec lesquels nous travaillons.

Organisation médicale :
La plateforme de Génétique Somatique s’appuie sur seize pathologistes moléculaires et une équipe technique spécialisée, qualifiée et habilitée en pathologie moléculaire.
Nous avons à cœur de développer des techniques de pointe, en accord avec les recommandations des sociétés savantes et au plus près des innovations technologiques.
En collaboration étroite avec le clinicien, nous participons au choix de la technique la plus adaptée pour chaque patient et à l’interprétation des résultats pour le choix stratégique du traitement dans des délais optimaux.
Le résultat de cette technique se fait sous le forme d’un compte-rendu intégrant l’ensemble des données pathologiques, diagnostiques, pronostiques et théranostiques, pour une prise en charge personnalisée du patient.

